从功能基因研究的角度来讲,相关的搜索,比如从序列数据库中,找同源的序列,或者对一个对一个新的基因功能进行鉴定,使用hmmer比使用blast有着更高的灵敏度已经更高的搜索速度,但其应用还远没有blast普及,这里是一篇入门级的介绍文章。
hmmer下载与安装
- 对于Mac OS/X, Linux, UNIX系统,用源代码编译安装:
# wget ftp://selab.janelia.org/pub/software/hmmer3/3.0/hmmer-3.0.tar.gz# tar zxf hmmer-3.0.tar.gz# cd hmmer-3.0# ./configure# make# make check
- 对于windows系统,直接下载二进制压缩包,解压就可以使用,下载地址:
http://hmmer.janelia.org/static/binaries/hmmer3.0_windows.zip
hmmer包含的程序
- phmmer: 与Blastp类似,使用一个蛋白质序列搜索蛋白质序列库;
phmmer tutorial/HBB HUMAN uniprot sprot.fa
- jackhmmer: 与psiBlast类似,蛋白质序列迭代搜索蛋白质序列库;
jackhmmer tutorial/HBB HUMAN uniprot sprot.fa
- hmmbuild: 用多重比对序列构建HMM模型;
- hmmsearch: 使用HMM模型搜索序列库;
- hmmscan: 使用序列搜索HMM库;
- hmmalign: 使用HMM为线索,构建多重比对序列;
hmmalign globins4.hmm tutorial/globins45.fa
- hmmconvert: 转换HMM格式
- hmmemit: 从HMM模型中,得到一个模式序列;
- hmmfetch: 通过名字或者接受号从HMM库中取回一个HMM模型;
- hmmpress:格式化HMM数据库,以便于hmmscan搜索使用;
- hmmstat: 显示HMM数据库的统计信息;
使用HMM模型搜索序列数据库
- 使用hmmbuild构建HMM模型,输入为Stockholm格式或者FASTA格式的多重比对序列文件(如:tutorial/globins4.sto),命令如下:
hmmbuild globins4.hmm tutorial/globins4.sto
globins4.hmm为输出的HMM模型
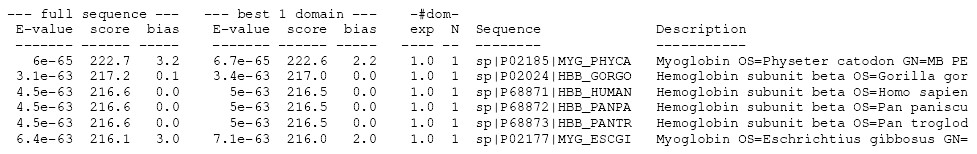
- 使用hmmsearch搜索蛋白质序列数据库,蛋白质序列数据库为FASTA格式,命令如下:
hmmsearch globins4.hmm uniprot sprot.fasta > globins4.out
globins4.out为输出的结果文件,如下:
使用蛋白质序列搜索HMM数据库
- 构建HMM数据库,HMM数据库是包含多个HMM模型的文件,可以从Pfam、SMART、TIGRFams下载,也可以自己由多重比对序列集中构建,如:
hmmbuild globins4.hmm tutorial/globins4.stohmmbuild fn3.hmm tutorial/fn3.stohmmbuild Pkinase.hmm tutorial/Pkinase.stocat globins4.hmm fn3.hmm Pkinase.hmm minifam
- 使用hmmpress格式化数据库,包括压缩以及创建索引,命令如下:
hmmpress minifam
这个步骤可以很快的执行完成,输出的内容如下:
Working… done.
Pressed and indexed 3 HMMs (3 names and 2 accessions).
Models pressed into binary file: minifam.h3m
SSI index for binary model file: minifam.h3i
Profiles (MSV part) pressed into: minifam.h3f
Profiles (remainder) pressed into: minifam.h3p
- 使用hmmscan搜索HMM数据库,命令如下:
hmmscan minifam tutorial/7LESS_DROME
输出如下:
这里主要是一个入门式的教程,介绍了hmmer的安装,以及最常用功能使用的命令示例。其他程序的使用,以及每个程序的详细参数说明,请参看官方手册,官方文档手册(pdf):ftp://selab.janelia.org/pub/software/hmmer3/3.0/Userguide.pdf