在使用Bowtie比对于完Chip-Seq的结果后,就需要用到MACS或者ERANGE来找出峰所在的位置了。但是由于ERANGE的设置比较复杂,所以最为流行的还是MACS。
我们首先来了解一下MACS的工作流程(http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2592715/),否则的话,许多参数都无法理解。
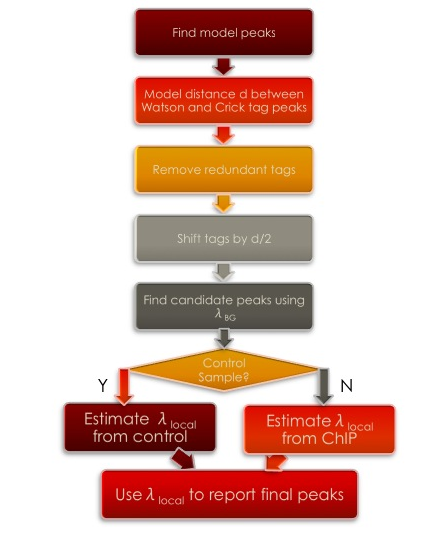
从这个工作流程中,我们可以看出,MACS首先的工作是要确定一个模型,这个模型最关键的参数就是峰宽d。这个d就是bw(band width),而它的一半就是shiftsize。而后就是依据峰高值来确定是否是一个峰。如果有对照的话,那么,对照当中也出现的峰就会被扣除。
我们现在来了解如何设置参数。
-t TFILE, –treatment=TFILE 输入文件名
-c CFILE, –control=CFILE 输入阴对文件名
-n NAME, –name=NAME 输入出文件名前缀
-f FORMAT, –format=FORMAT 输入文件格式,默认值为AUTO,可选的值为”BEG”,”ELAND”,”ELANDMULTI”,”ELANDMULTIPER”,”ELANDEXPORT”,”SAM”,”BAM”,”BOWTIE”等。
-g GSIZE, –gsize=GSIZE 比对模板大小。格式可以是:1.0e+9,或者1000000000,也可以缩写:’hs’ for 人类 (2.7e9), ‘mm’ for 大鼠(1.87e9), ‘ce’ for 线虫 (9e7) and ‘dm’ for 果蝇 (1.2e8), 默认值:hs
-s TSIZE, –tsize=TSIZE 设置为短序列的长度,默认值为25
-p PVALE, –pvalue=PVALUE 非峰可能性截取值,默认值为1e-5,这个值不能大太,超过0.9的话,可能无法输出正确的结果
-m MFOLD, –mfold=MFOLD 峰值高度相对于本底的比值,默认值为10,30。也就是说,最低值不能少于10,但比值超过30也不认为它是正常的一个峰。一般而言,低值设置为10是一个很好的区分点。如果这个值还是无法得到满意的结果,那么可以设置得更低,但最好还是使用–nomodel参数,使–nomodel设置为True,然后再传递–shiftsize及–bw参数给MACS。–shiftsize默认值为100,而–bw的默认值为300。
–diag 生成完整报表,会包括是否为真峰的可能性,但会严重拖累运算速度。
例:
- macs –gsize=hs –tsize=28 –format=BOWTIE –name=Pol2 –diag –mfold=5,50 -t Pol2ChIP.map -c Pol2ChIP.control.map
原文来自:http://pgfe.umassmed.edu/ou/archives/2288
参见更多:http://liulab.dfci.harvard.edu/MACS/00README.html



1F
你好,我想请问一下,使用MACS寻找峰值的原始数据应该是怎样的呢,我是双末端测序的bed数据,然后我要把数据变成怎样的文件才能进行MACS寻找峰值的工作呢?
2F
bed文件可直接call peak
来自外部的引用