1、首先安装Aspera
参考《使用Aspera从EBI或NCBI下载基因组数据》,保证该软件在服务器上已经可用。
测试代码,注意最后有个点,代表下载到当前目录下:
- ascp -i /your-path-to/.aspera/connect/etc/asperaweb_id_dsa.putty anonftp@ftp-private.ncbi.nlm.nih.gov:/sra/sra-instant/reads/ByStudy/sra/SRP/SRP021/SRP021493/SRR834864/SRR834864.sra
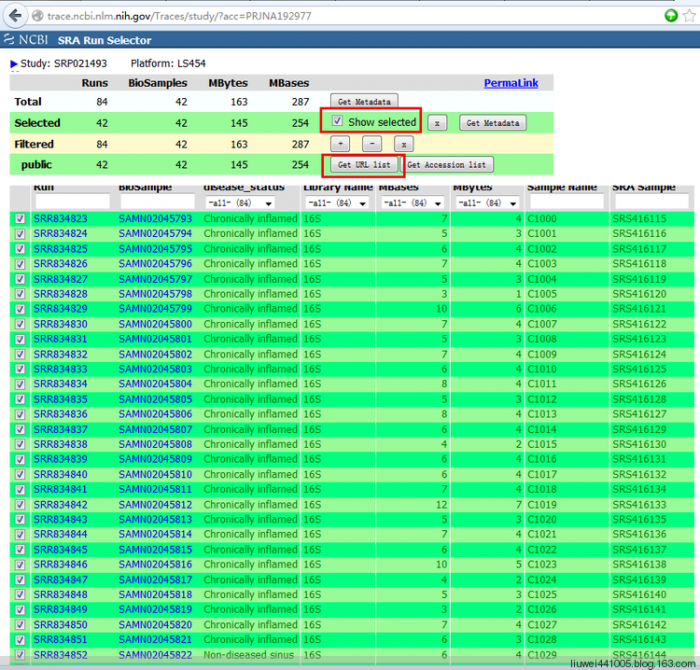
2、参考《Viewing and downloading tabular metadata with the SRA Run Selector》下载宏基因组数据对应的下载链接
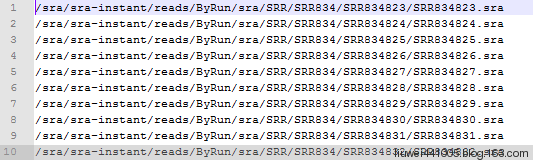
下载后得到的链接地址如下:
3、开始下载:
- ascp -i /your-path-to/.aspera/connect/etc/asperaweb_id_dsa.putty --mode recv --host ftp-private.ncbi.nlm.nih.gov --user anonftp --file-list SRR_Download_List.txt
4、用SRA tools把SRA格式转换成fastq
下载tools https://www.ncbi.nlm.nih.gov/books/NBK158900/,linux版本,下载解压后可以直接使用,添加个环境变量即可。
命令:
- fastq-dump.2.3.5.2 -A SRR*.sra
5、OK,大功告成!
my test command:
- ascp -i /share/home/jialj/.aspera/connect/etc/asperaweb_id_dsa.putty --mode recv --host ftp-private.ncbi.nlm.nih.gov --user anonftp --file-list SRR_Download_List_file_list.txt
原文来自:http://liuwei441005.blog.163.com/blog/static/13570581120144935013905




1F
为什么现在需要输入密码了呀?