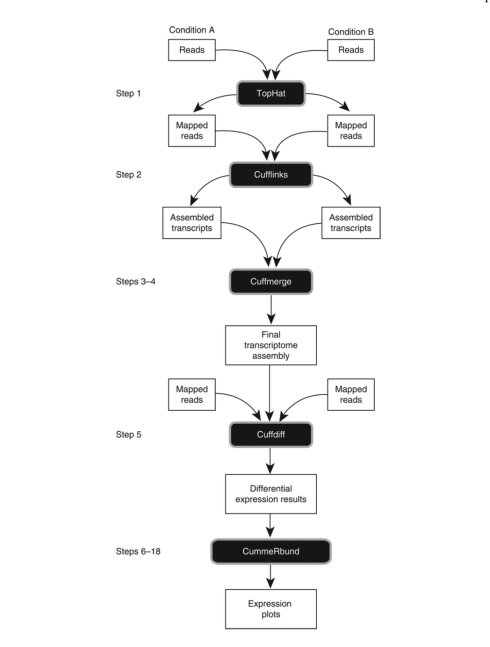
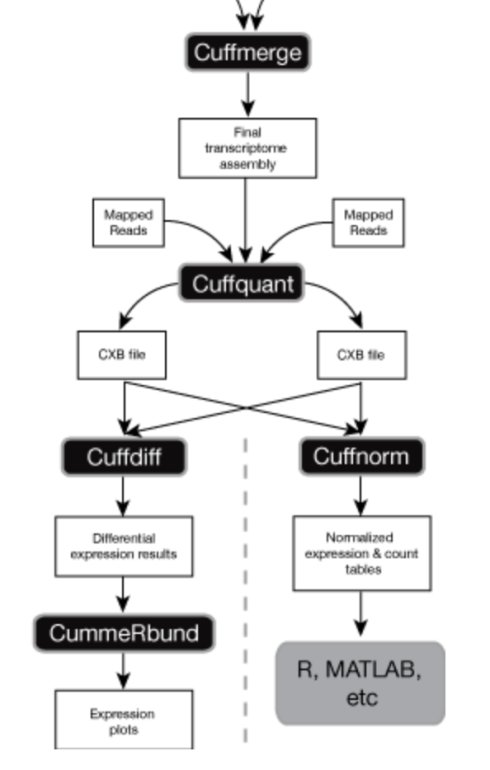
整个分析用TopHat进行比对,比对完成后将比对输出作为cufflinke拼接的输入(单独拼接),将单独拼接的结果使用cuffmerge混合,然后使用cuffdiff做差异,使用r软件包CummeRbund输出差异比大的相关图形。(目前已经改版有点变化)。
现在改动的部分:
Tophat实际上就是bowtie变体,只不过bowtie只能比对DNA,tophat可以比对RNA,简单就是剪掉过内含子的序列。主要是针对可变剪切的比对,用于RNAseq定量分析,一般存在参考的注释信息。
Tophat网址:http://ccb.jhu.edu/software/tophat/index.shtml
需要事先准备的文件有:
参考基因组的fasta文件以及用bowtie建立的index文件
测序数据(fastq)
参考的转录本注释信息(gtf格式)
Usage:
tophat [options]* [reads1_2,...readsN_2]
for example:
tophat -p 8 -G genes.gtf -o C1_R1_thout genome C1_R1_1.fq C1_R1_2.fq
-p 代表线程
-G 代表转录本注释信息
-o 输出文件夹
--segment-length 25 (将redas分成的最小比对片段)
--segment-mismatches 1 (片段比对错配碱基数)
--library-type (是否链特异性)fr-unstranded
—transcriptome-index (转录本的bowtie-index文件)
比对输出文件:
accepted_hits.bam(比对输出)
junctions.bed
insertions.bed and deletions.bed
################
cufflinks拼接
Usage: cufflinks [options]*
for example:
cufflinks -p 8 -o C1_R1_clout C1_R1_thout/accepted_hits.bam
cufflinks -p 8 -o C1_R2_clout C1_R2_thout/accepted_hits.bam
cufflinks -p 8 -o C1_R3_clout C1_R3_thout/accepted_hits.bam
cufflinks -p 8 -o C2_R1_clout C2_R1_thout/accepted_hits.bam
cufflinks -p 8 -o C2_R2_clout C2_R2_thout/accepted_hits.bam
cufflinks -p 8 -o C2_R3_clout C2_R3_thout/accepted_hits.bam
建立一个文件命名为:assemblies.txt,assemblies.txt内容如下:
./C1_R1_clout/transcripts.gtf
./C2_R2_clout/transcripts.gtf
./C1_R2_clout/transcripts.gtf
./C2_R1_clout/transcripts.gtf
./C1_R3_clout/transcripts.gtf
./C2_R3_clout/transcripts.gtf
运行Cuffmerge:
- cuffmerge -g genes.gtf -s genome.fa -p 8 assemblies.txt -o /merged.gtf
输出产生一个GTF文件:merged.gtf
运行Cuffquant:
Usage:
cuffquant [options]*
参数:-o/—output-dir 输出目录,默认为当前目录
输出文件:abundances.cxb
运行Cuffdiff:(计算每个样本基因、转录本的FPKM)
组与组比较:
cuffdiff --use-sample-sheet
sample_sheet.txt内容如下:
sample_id group_label
C1_R1.sam C1
C1_R2.sam C1
C2_R1.sam C2
C2_R2.sam C2
如果两个条件之间比较:-C
condition_A condition_B
Ctrl Mutant_X
Ctrl Mutant_Y
Ctrl Mutant_Z
名字的命名至少匹配样本名称
以前版本的命令:
cuffdiff -o diff_out -b genome.fa -p 8 –L C1,C2 -u merged_asm/merged.gtf
./C1_R1_thout/accepted_hits.bam, ./C1_R2_thout/accepted_hits.bam,
./C1_R3_thout/ accepted_hits.bam
./C2_R1_thout/accepted_hits.bam,./C2_R3_thout/accepted_hits.bam,./C2_R2_thout/ accepted_hits.bam
(重复用逗号隔开)
原文来自:http://blog.sina.com.cn/s/blog_83f77c940102v7wl.html