一:下载安装该软件
软件:NCBI提供的igblastn(linux环境)
需要自己去NCBI的ftp里面下载
ftp://ftp.ncbi.nlm.nih.gov/blast/executables/igblast/release/
要保证igblastn程序文件和以下三个文件夹在同一目录,可以自行下载ncbi的igblast程序,同时要下载这些东西。

其中database根据物种需要自行选择或者自己建库
二:准备数据
输入文件:各个样本经以上步骤处理好的fasta文件
输出文件:得到blast比对结果,一般处理后文件会增大10倍以上
三:运行命令
软件命令:对不同的物种需要不同的库文件,不同的BCR,TCR需要不同的比对策略,好好看说明书
对BCR 对TCR


处理结果如下,每个文件约2.5G大小
我一般是好几个样本一起跑,两百兆的文件需要跑四个小时左右!批处理命令如下,反正一般人也看不懂,就忒给有需求的人!
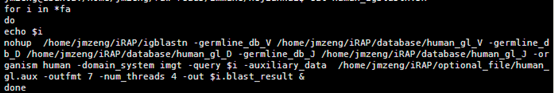
四:输出文件解读
这个就复杂了,一篇文章根本解释不清楚呀
在我写这篇日志的时候,我后台运行的igblastn程序还在运行,估计明天早上才能跑完
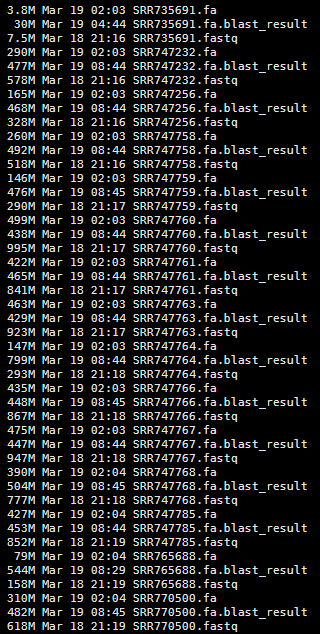
这是我下载的一篇文献里面的数据,可以看到每个fa文件都输出了很大的blast result文件,需要打开一个个解读。
原文来自:http://www.bio-info-trainee.com/352.html


