一、下载及安装软件
这个软件需要edu邮箱注册才能下载,可能是仅对科研高校开放吧。所以软件地址我就不列了。
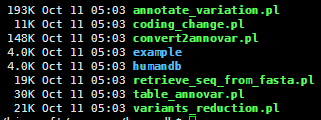
它其实是几个perl程序,比较重要的是这个人类的数据库,snp注释必须的。
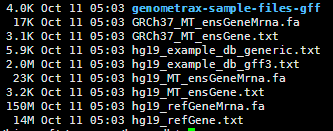
参考:http://annovar.readthedocs.org/en/latest/misc/accessory/
二,准备数据
既然是注释,那当然要有数据库啦!数据库倒是有下载地址
http://www.openbioinformatics.org/annovar/download/hg19_ALL.sites.2010_11.txt.gz
也可以用命令来下载
- Perl ./annotate_variation.pl -downdb -buildver hg19 -webfrom annovar refGene humandb/
然后我们是对snp-calling流程跑出来的VCF文件进行注释,所以必须要有自己的VCF文件,VCF格式详解见本博客另一篇文章,或者搜索也行
http://vcftools.sourceforge.net/man_latest.html
三、运行的命令
首先把vcf格式文件,转换成空格分隔格式文件,自己写脚本也很好弄
- perl convert2annovar.pl -format vcf
- /home/jmzeng/raw-reads/whole-exon/snp-calling/tmp1.vcf >annovar.input
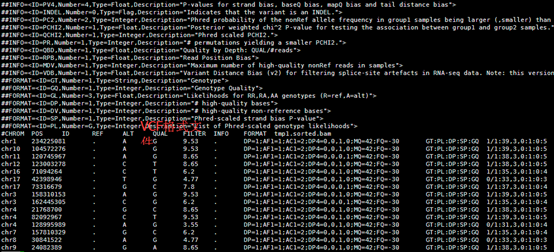
变成了空格分隔的文件
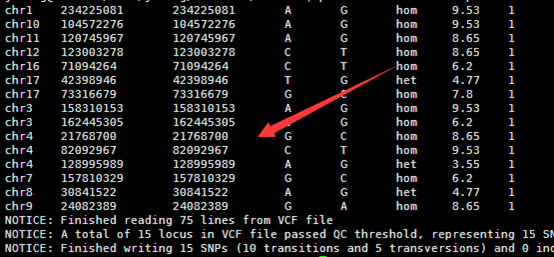
然后把转换好的数据进行注释即可
- ./annotate_variation.pl -out ex1 -build hg19 example/ex1.avinput humandb/
四,输出文件解读

原文来自:http://www.bio-info-trainee.com/441.html


