肖景发, 于军
中国科学院北京基因组研究所, 中国科学院“基因组科学及信息”重点实验室
摘要
根据DNA核苷酸组分的动态变化规律将遗传密码的传统排列按密码子对GC和嘌呤含量的敏感性进行了重排. 新密码表可划分为两个半区(或1/2区)和四个四分区(或1/4区). 就原核生物基因组而言, 当GC含量增加时, 物种蛋白质组所含的氨基酸倾向于使用GC富集区和嘌呤不敏感半区所编码的氨基酸, 它们均使用四重简并密码, 对DNA序列的突变具有相对鲁棒性(Robustness). 当GC含量降低时, 大多数密码子处于AU富集区和嘌呤敏感半区, 这个区域编码的氨基酸具有物理化学性质的多样性. 因为当密码子第三位核苷酸(CP3)在嘌呤和嘧啶之间发生转换时, 密码子所编码的氨基酸也倾向于发生变化.
关于遗传密码的进化存在多种假说, 包括凝固事件假说、共进化假说和立体化学假说等, 每种假说均试图解释遗传密码所表现出来的某些化学和生物学规律. 基于遗传密码的物理化学性质、基因组变异的规律和相关的生物学假说, 我们提出了遗传密码分步进化假说(The Stepwise Evolution Hypothesis for the Genetic Code). 在人们推断的最原始的RNA世界里, 原初(Primordial)遗传密码从只能识别嘌呤和嘧啶开始, 编码一个或两个简单而功能明确的氨基酸. 由于胞嘧啶C的化学不稳定性, 最初形成的遗传密码应该仅仅由腺嘌呤A和尿嘧啶U来编码, 却可得到一组七个多元化的氨基酸. 随着生命复杂性的增加, 鸟嘌呤G从主载操作信号的功能中释放出来, 再伴随着C的引入, 使遗传密码逐步扩展到12、15和20个氨基酸, 最终完成全部进化步骤.
遗传密码的进化过程同时也伴随以蛋白质为主体的分子机制和细胞过程的进化, 包括氨酰tRNA合成酶(AARS)从初始翻译机器上的脱离、DNA作为信息载体而取代RNA以及AARS和tRNA共进化等基本过程. 分子机制和细胞过程是生命的基本组成元件, 它们不但自己不断地趋于完善, 也促使生命体走着不尽相同的道路, 要么维持鲁棒性(Robustness, 如细菌), 要么寻觅多元化(Diversity, 如节肢动物和植物), 要么追求综合性(Complexity, 如脊椎动物).
自从密码子被全部发现以来, Crick[1]就将遗传密码表排列成化学家所认可的形式. 尽管后来有些特殊表现形式的列方式(如同心圆、八卦式和二元密码等), 但其基本排布一直延续至今[1~3]. 遗传密码以4个脱氧核糖核苷酸作为基本符号来组成遗传信息, 并以20个氨基酸作为基本结构单元来构建蛋白质. 遗传密码是使用4个碱基(两个嘌呤: 腺嘌呤A和鸟嘌呤G; 2个嘧啶: 尿嘧啶U和胞嘧啶C)构成的三联体密码子, 共64个, 分别对应20个氨基酸或翻译起始和终止信号. 生物体要将DNA分子中储存的信息内涵转变成功能内涵, 就要利用信使mRNA、解码分子tRNA和完整翻译机器等多重功能. 各种复杂分子机制和细胞过程的诞生和成熟一定会反映生命从RNA世界到RNA-蛋白质世界, 再到RNA-蛋白质-DNA世界逐渐转变的过程, 遗传密码作为一个独立的生物学机制也一定是漫长生命进化过程中的一个必然产物.
20世纪60年代初, 实验分子生物学最大的进展就是解码遗传密码, 发现它在生命有机体中, 基本是统一的. 自此不同的假设均试图解释遗传密码的信息和化学特性, 从简单的凝固事件假说到更复杂的统计学、共进化和立体化学理论. 凝固事件假说认为密码子与氨基酸的对应关系是在某个生命发生时段里被固定下来, 并且很难被改变[2], 这个假说一直被基于适应性、历史性和化学性的不同论点所挑战[4]. 尽管关于遗传密码的进化也有人提出过不同的假设, 但是解释密码子的分配原则、物理化学性质的相关性和DNA组分变化对密码子使用频率的牵动, 从而揭示遗传密码表的生物学本质仍然是一个不小的挑战[2,5].
1 重排遗传密码表
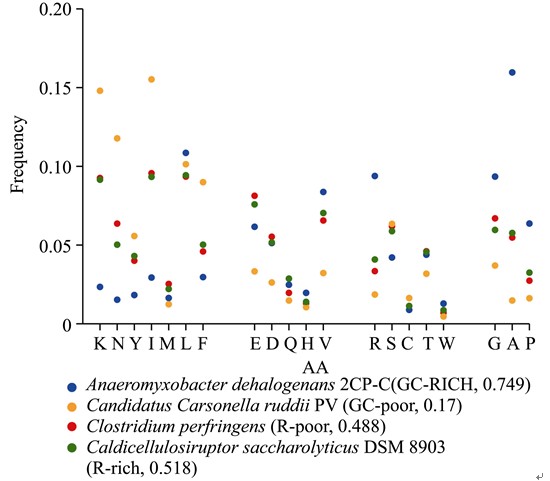
重排遗传密码表有3个重要原因. 首先, DNA序列有4个最基本的可度量的变化, 即核苷酸序列、序列长度、GC含量和嘌呤(R或AG)含量. 假如把核苷酸序列和长度相对于时间的变化暂时不考虑, 那么只有后面的两个变量对于传统的遗传密码表具有影响力, 所以重排应该以GC和嘌呤含量的变化为主线. 但以前大家熟知的密码表排列只是为了简明和清晰地显示密码子和氨基酸的一一对应关系, 却忽略了密码表本身对氨基酸物理化学性质多样性的表现和DNA编码承受突变的鲁棒性等明显信息. 因此, 有必要把传统的密码表进行重新排列[6], 使其能够表现信息内涵和功能内涵之间的基本关系. 其次, 当GC含量和嘌呤含量变化时, 希望从密码表中找出相应蛋白质组成变化的线索. 图1展示了GC含量和嘌呤含量在极端状态下4个微生物基因组的氨基酸组分分布.
图1 在GC含量和嘌呤含量极端状态下4个微生物基因组的氨基酸组分分布
1.1 密码表的分区和基本内涵
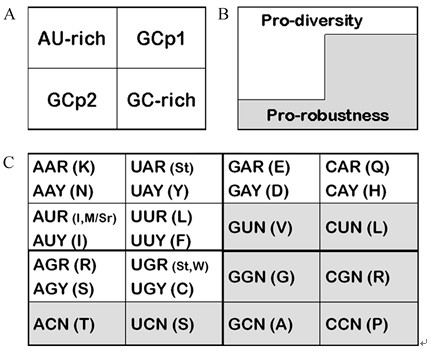
遗传密码表可按密码子对于GC含量变化的敏感性分为4个四分区(或称1/4区, 见图2): AU富集、GC富集和两个GC不敏感区(GCP1和GCP2区). 如果暂时忽略密码子第三位的变化, 可以看出AU和GC富集区对GC含量变化非常敏感. 还可以根据密码子在这个区里的分布预测密码子第二位的GC含量大于第一位的GC含量(GC2>GC1), 因为GCP2区里含有终止密码子, 尽管密码子使用偏好可能使真实统计变得复杂些. 在AU富集区里编码的氨基酸具有理化性质的多元性, 共有16个密码子. 除了编码7个氨基酸外, 还编码两个终止密码子和一个起始密码子. 比较而言, GCP1和GCP2区分别只编码6个氨基酸, 而GC富集区则只编码4个氨基酸. AU富集区只占编码总量的1/4, 却含有足够多的信息, 使得很容易推测这个区可能在生命的早期(只有A和U存在时)作为原始的遗传密码来编码简单的蛋白质组分或更简单的多肽. 正是这些简单的蛋白质使原始生命体的分子机制和细胞过程开始变得复杂和多元.
新表按照对嘌呤含量变化的敏感性可划分成两个半区. 同时, 也可按照编码氨基酸在三联密码子第三位(CP3位)嘌呤和嘧啶(Y)间的变化再分为四重简并和两重简并(两个特例为AUR和UGR). 基于它们对所编码氨基酸功能的暗示, 将这两个半区分别叫做趋变半区(Pro-diversity)和趋棒半区(Pro-robustness), 简称为PD半区和PR半区. 这种清晰的划分使人们能进一步观察、分析和预测DNA序列变化与密码子(氨基酸)的关系. 首先值得关注的是具有六联码(六重简并)的3个氨基酸: 丝氨酸(Ser)、精氨酸(Arg)和亮氨酸(Leu). 它们的四联部分和二联部分恰好都分布在两个不同的半区里, 因此它们可以通过内部密码子间的转换分别平衡在所跨的半区里的分布(图2(C)). 因为它们的简并性, 可以预测它们是所有物种中相对最富集的氨基酸. 该分布也可以用这3个氨基酸的特殊物理化学性质(如重要催化残基)和独特功能域骨架(如Leucine zipper和SR富集功能域等)在细胞蛋白质组中的丰度来解释. 另外, 所有的无义密码子均分布在PD半区. 当GC含量变化时, UAA, UAG和UGA会转变成其他氨基酸, 具有在3′末端扩展蛋白质长度的潜力. 另外, 由于分布在密码表多个区域, 两个碱性氨基酸Arg和赖氨酸(Lys)对GC含量变化具有鲁棒性. 而两个酸性氨基酸, 天门冬氨酸(Asp)和谷氨酸(Glu), 同时分布在GCP2区对GC含量变化不敏感, 同样可以预测它们在蛋白质的中丰度会较高, 同时两者的化学性质具有很高的相似性, 成为典型的伪四联码(Pseudoquartet). 最后, 脯氨酸(Pro)处于GC富集区的角落处, 只有当GC含量通常较高时才会在蛋白质组中多见, 用它的肽键所形成的小拐角来取代多个氨基酸所形成的大拐角. 总之, 在DNA组分变化时, 20种氨基酸也随之发生有规律的变化, 造成蛋白质组(几乎所有的蛋白质)的鲁棒性和多元化, 这些互动和变化的规律一定受限于氨基酸的物理化学性质和蛋白质的生物功能, 最后形成了一种相对稳定的关系, 这就是密码表所要体现的关系.
图2 重新排布的遗传密码表和按照GC含量和AG含量遗传密码表的两种划分方式
R: 嘌呤; Y: 嘧啶; St: 终止密码子; Sr: 起始密码子
1.2 遗传密码形成的基本要素
重新排布的密码表揭示了密码子的分布与GC含量变化的直接关系(真细菌基因组的GC含量的波动幅度在20%~80%之间). 首先以GC含量变化作为初始参数, 可以把遗传密码分成两部分. 其次, 高GC含量的遗传密码多为四联码, 它们的形成是为了缓解GC含量增加的影响(CP3位), 减轻DNA突变压力, 因为GC富集区编码的氨基酸很少参与催化活性以及起始和终止信号. 另外, 物理化学性质多样性的氨基酸和信号密码子大多集中在AU富集区. 同时, GC不敏感区的功能主要是保护功能丰富性氨基酸, 而最原始的理化性质往往已经被其他分布所平衡, 例如亮氨酸(Leu)和丙氨酸(Ala)已经在AU富集区和GC富集区间造成疏水性氨基酸间的平衡.
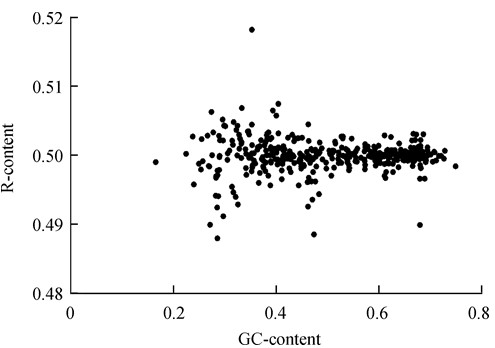
嘌呤含量变化是第二个需要考虑的因素. 其实真细菌基因组嘌呤含量的波动在40%~60%之间, 并非Chargaff规则所说的50%[7,8]. 图3展示了真细菌基因组GC含量和嘌呤含量变化的基本关系. 根据对嘌呤含量的敏感性将密码表划分PD和PR两个半区, 这种划分清晰地把四重简并密码和其他密码划分开来. 趋棒半区的5个氨基酸(六重简并的除外)具有低多样性和高突变稳定性的特点, 每一个氨基酸都在表中其他位置有物理化学性质相近的其他氨基酸, 变化仅仅体现在某些细微的程度上(Subtlety).
重排密码表的另一个显著特点是把那些体积最小但理化性质特异的氨基酸分布在DNA组分变化时不大容易发生的区域(变化均为颠换而不是转换). 假如按照大小作为参数排列这4个最小的氨基酸, 甘氨酸(Gly)(侧链体积60.1和表面积75), Ala(88.6和115), Ser(89和115)和半胱氨酸Cys(108.5和135), 其他氨基酸都比这4个氨基酸大.
图 3 真细菌基因组GC含量和嘌呤含量变化的基本关系
1.3 密码子合理分布的最终目的是平衡蛋白质组分的多样性和对DNA突变的鲁棒性
遗传密码分布的实质是体现一种平衡, 一种既可利用DNA突变来增加蛋白质组的多样性又可利用密码子的分布来减轻突变所造成损失的能力. 尽管遗传密码表可划分GC与嘌呤的敏感性和不敏感性、氨基酸多样性和简并性、突变敏感性和突变耐受性, 但是这些划分并没有任何明显的倾向性. 可以预见这个平衡有时也会遭到严重破坏, 比如GC含量到极至和真细菌的嘌呤含量也超过Chargaff规则时[9], 这种压力就会毫无保留地体现在蛋白质序列的改变上. 但在密码表嘌呤敏感(PD和趋变)半区的氨基酸之间也具有某些补救性的分布. 例如Asp和Glu就位于同一个四分区, 当只有负电荷变得重要(大小和容积不太要求)时, 嘌呤和嘧啶在CP3位置变化的影响就被弱化了. 扩展来说, 在PD半区有几种相似的情况出现, 包括Q/H(尺度)、M/I(疏水性)、L/F(疏水性)、R/S(极性)、W/C(极性)和K/N(极性)(表1). 这种分析体现了很强的合理性, 目前还没有发现在同一分组(四联位)中编码的氨基酸具有混合特征, 如: 疏水性对亲水性, 极性对非极性等. 因此, 密码表的这些基本排布原则, 称之为“最小损伤原则”(Minimal Damage Principle).
2 关于遗传密码起源的种种假说和新的分步进化假说
关于密码子的起源和进化一直是科学界广泛探讨的一个重要话题, 多年来的主要研究进展包括凝固事件假说、共进化假说和立体化学假说等[10~19], 以及本研究组最近提出的关于密码分步进化的新推测[20,21].
2.1 共进化假说
共进化假说(Co-evolution Hypothesis)提出标准密码(Canonical Code)是从早期原始的简单密码进化而来的. 这个原始的密码可能是由64个密码子通过高度简并, 只编码少量的氨基酸. 现在的氨基酸密码可能是通过篡夺和它具有相近生物合成路径氨基酸的密码而得到的[10~14].
L. Klipcan[13]提出假设氨酰tRNA合成酶(AARS)和氨基酸及其辅因子的生物合成存在关联性, 并指出Class II 的AARS 相对应的氨基酸较早出现, 而Class I 的AARS对应的氨基酸在进化上出现较晚. 他还指出最原始的遗传密码可能是5个基本氨基酸Ala, Gly, Ser, Asp, Glu和缬氨酸(Val). 所有这些氨基酸均是GC富集和相应的生物合成路径最短和最简单的氨基酸, 以及相应的AARS属于Class II. 接着引入4~5个氨基酸对应遗传密码的扩展, 分别为天门冬酰胺(Asn)、苏氨酸(Thr)、Pro和谷氨酰胺(Gln). 另一个可能是Arg, 这些氨基酸生物合成路径的复杂性基本处于中间位置. 第三阶段所有编码的氨基酸均被引入, 这次引入的氨基酸和Class I 的AARS有很强的相关性, 同时这些氨基酸使用的遗传密码多含尿嘧啶, 最后引入的氨基酸合成通常需要非常长的代谢途径.
B.K. Davis[14]通过氨基酸生物合成路径长度和其他关于密码分配时间分子描述符比较来检验和构建密码的进化的主要阶段, 认为密码树来源于16个三联体密码(NAN系列)对应于4个固N氨基酸 (Asp, Glu, Asn, Gln). 通过这些氨基酸的寡聚化形成包含多聚阴离子氨基酸的肽链把不带电荷氨基酸定位于带正电荷的矿物质表面. 为了降低基因突变导致基因编码不可解读的危险性,遗传密码需要进一步扩展. 这一步通常起始于翻译序列5'端密码, 对应疏水性氨基酸NUN系列密码分配相对较慢, 它编码大部分非极性氨基酸. 最后带正电荷和芳香性氨基酸最后被引入, 自此具有酸碱催化功能的酶的合成也成为可能, 对应的两种类型氨酰tRNA合成酶应该出现在这一时期.
2.2 立体化学假说
立体化学假说指出密码子的分配直接与RNA和氨基酸之间的化学作用密切相关, 因此密码的立体化学本质扩展到氨基酸和相应的密码子之间的物理和化学方面的互补性[15~19].
Wu[17]提出一个简单密码进化的新假设, 基于翻译过程中每一步3个碱基的读码机制包含从单体到双联体密码再到三联体密码子的进化过程. 建议三联体密码是从两种类型双联体密码逐渐进化得来的, 这两种双联体密码划分是按照三联体密码中固定的碱基位置划分的, 包括前缀密码子(Prefix codons)和后缀密码子(Suffix codons). 这个假设可以解释遗传密码多种特征, 如其中包含四重简并三联体密码和两重简并三联体密码的模式, 翻译错误最小化性质的本质和为什么只有20个编码的氨基酸.
Delarue[18]按照tRNA的氨酰基化机制把密码子的分配表当成一个典型的反对称分布模式, 这种模式可以通过连续的二元决定阈逐步降低密码子的模糊性, 可以通过定义一个简单的规则, 使每个密码子的解码可以按照二元分类方式, 从而提供相应密码和密钥. 按照模型密码子的最终分化步骤包括: (1) Y/R 在密码子的第二个位置打破对称性分配; (2) 在密码子的第二个位置按照 G/A 和 C/U特殊性; (3) R/Y在密码子的第一个碱基位置对称性打破; (4) 在密码子的第一个碱基位置对称打破A/G和C/U; (5) Y/R在密码子的第三个位置按照对称性打破.
按照分子识别理论,通过无义密码和有义密码矩阵、反义和反无义密码矩阵以及与疏水性矩阵的比较, 可以明显地把氨基酸分成两个组: 疏水性和亲水性组, 这个划分直接与蛋白质的折叠相关. 初始的遗传密码只区分这两种类型的氨基酸, 使蛋白质具有折叠成稳定三维结构的能力, 这种多肽与RNA分子形成核糖体蛋白复合物来稳定编码RNA. Houen[19]指出, 基于上述推断原始的密码子包括Leu, Arg和Ser. 遗传密码的进一步扩展增加功能性亲水氨基酸, 因为暂时还没有增加结构疏水性氨基酸的必要性. 在遗传密码进化的某一阶段, 双链DNA作为遗传信息的载体取代RNA, 此过程需要高度特异性的酶体系, 因此需要对遗传密码进行进一步优化而引入其他氨基酸.
2.3 遗传密码的分步进化假说
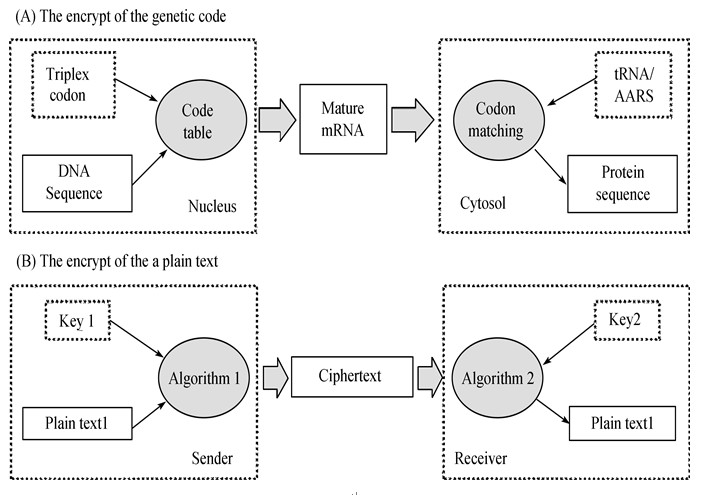
基因密码与人造密码具有相似性, 即需要成对的算法和两个相应的解码钥匙共同创造和完成解码工作, 基因密码的算法和钥匙的组成材料是由两个细胞空间的成分组成: 细胞核和细胞质. 细胞核的成分是DNA(Text1)和三重密码子(Key1)共同组成遗传密码(Algorithm1), 胞质里的成分包含蛋白质(Text2)和mRNA-ribosomes-tRNA 组成的复合体(Algorithm2), 通过氨酰tRNA合成酶(Key2)进行解码, 使mRNA序列所承载的编码内容被解码(图4). 这个过程是通过长期进化演变出来的分子机制和细胞过程统一实施的.
图 4 遗传密码(A)和文本密码(B)的比较
(1) RNA世界和早期遗传密码. RNA世界的存在首先被RNA分子具有相应催化功能的生物学特性所支持[22~26]. 在RNA世界里, RNA具有双重的功能, 既是信息载体也是功能载体. 因为生命的基本分子机制和细胞过程起源于RNA世界, 所以没有理由说遗传密码不起源于RNA世界. 在RNA世界里RNA分子可以组成简单的核苷酸多聚物, 这种多聚物在近亿年的成熟期里, 为生命提供了足够的功能上的复杂性和多样性. 原始细胞可以通过相互争斗和吞噬获得基本的组成成分,因此基于模板的RNA合成可能对于生命的初始不是必需的. 可以想象这些RNA分子可以通过简单的聚合酶来合成, 通过自身剪接或化学修饰转变为其他相似的结构, 从而达到结构的可变性和功能的多样性. 此外, RNA的编辑(RNA Editing)也一定起了非常重要的作用, 这一分子机制一直延续到现在, 在包括人类在内的高级物种中仍然存在.
在现代生物世界里, 剪接体(Spliceosome)通常是用于RNA分子的剪接, 由蛋白质和RNA分子组成. 可以做两个假设, 生命可能起源于类真核有机体的原型细胞(在DNA引入之前)而不是类原核有机体的原型细胞. 在RNA组成的翻译机器(Translational Machinery)没有形成之前, 初始遗传密码可能不是必需的. 一旦这个初始生命进入到RNA-蛋白质组成的世界时, 多肽才逐渐按照密码子开始有序合成, 遗传密码就开始发挥其作用了. 可以认为有序的生命可以在与相对无序生命的争斗中更容易获胜和取得繁衍的空间.
现在可以推测初始遗传密码在RNA世界存在和进化的基本过程和起源时的基本逻辑关系. 初始生命一定比较简单, 分子间相互作用也比较宽松, 最小的编码系统可能只要区分嘌呤R和嘧啶Y就够了. 假定现代密码在生命的早期阶段已经被统一并相对忠实地继承了RNA密码的基本关系, 这个可能的原始编码就至少有7个氨基酸(I和M视为等同; 图5), 同时也有起始和终止密码子. 这7个氨基酸的侧链具有广泛的物理化学性质(氨基、酰基、苯环、羟基、酚基、烃链和甲硫基等), 但是没有小的和酸性的氨基酸. 可以推测: 体积小的氨基酸在初始蛋白质相互作用中的作用显然不如大的重要, 而碱性氨基酸的功能对于酸性DNA则是显而易见的. 另外的一种可能性是氨基酸与tRNA以及AARS之间的关系不是十分明确, 一个密码子对应多氨基酸的情况可能在遗传密码成熟前是普遍存在的[27]. 由于7个氨基酸的编码区处于现代密码表的AU富集区, 可以确信初始密码子始于这个区域, 后来扩展到嘌呤敏感区即所谓趋变半区. 这个阶段的存在既复合由简到繁的逻辑, 也迎合了实验的证据, 那就是C的不稳定性和G在RNA操作功能上的作用[21, 28~29].
图 5 遗传密码的分步进化假说
最初形成的遗传密码应该仅仅由腺嘌呤A和尿嘧啶U来编码, 共编码7个多元化的氨基酸, 随着生命复杂性的增加, 鸟嘌呤G从主载操作信号的功能中释放出来, 再伴随着C的引入, 使遗传密码逐步扩展到12, 15和20个氨基酸
因为初始密码的核心作用, 有必要再拓展一下讨论. 认为初始密码的简单化是由初始翻译机器的原始性来决定的. 第一个AARS可能首先被作为蛋白质加工机器的组成部分(如核糖体的亚基之一), 它可以: (1) 不区别氨基酸的微妙差别, 或只识别简单的部分; (2) 将氨基酸底物按核糖体的排列随机加在肽链上; (3) 与氨基酸的代谢途径耦联, 受底物浓度的影响. 显然, 这个部分的分离是蛋白质复杂性增加的关键, 随后AARS不得不从蛋白质合成机器上分离出来, 对底物的专一性产生并不断增加, tRNAs也随之远离核糖体. 由于初始RNA编码的多肽通常用于细胞的完整性和对遗传物质的保护, 所以可以确信蛋白质的初始功能是与RNA结合和稳定膜的结构, 因此初始氨基酸必然是碱性、芳香性和疏水性的氨基酸. AARS的分离和独立进化是为了保证蛋白质成分的多样性, 也就是功能的多样性. AARS的进化树进一步成为本研究推断的佐证[30,31]. 比如, AARS必须区分两个极性氨基酸Asn和酪氨酸(Tyr)、两个芳香性氨基酸Phe和Tyr, 但是对于Leu, Ile和甲硫氨酸(Met)则没有必要区分, 因此AARS两个家族的分化和本推论是完全一致的.
(2) 遗传密码的第一次拓展. 对于遗传密码的延伸, 引入新的构建元件, 本文有两个基本假定: 鸟嘌呤G的引入和A(腺嘌呤)-I(次黄嘌呤)编辑机制的作用, 这两种假设给原始的mRNA提供了显著的结构多样性和编码能力. 尽管这两种假设并不互相排斥, 两者之间可能独立或同时存在, 只是为了简单化才把它们分别进行讨论. 首先, G引入到转录本中存在有限的途径(同时作为蛋白质合成的能源和过程信号), 由于AG和GU作为剪切体的识别信号, 因此密码子的扩展只限于色氨酸(Trp), Glu, Asp, Cys和Gly. 这些扩展的氨基酸彼此之间具有显著不同的侧链理化性质: Trp是最大的氨基酸, Asp和Glu是带负电荷的氨基酸, Cys是可形成二硫键的氨基酸, Gly是最小的氨基酸. Glu, Asp和Cys的出现为蛋白质自身结构的稳定性和独立性提供了必要的结构单元. 其次, A-I的可选择性和经常性的RNA编辑使mRNA的复杂性不断增加. A和I的同时存在, 使密码子扩展到更多氨基酰-tRNA. 这个推断得到AARS分布的支持, 氨基酸的拓展和AARS家族的分类具有同步的规则, 而且核苷酸的修饰作用也被后续的生命机制所延续, 如tRNA反密码子和mRNA的摆动机制.
(3) 遗传密码的第二次拓展. 当GU和AG从作为剪接信号功能释放出来以后(剪接体的结构和功能随着蛋白质的演变而复杂化和精密化), 遗传密码引入了Arg, Ser和Val. 氨基酸的个数变成15个, 这次扩展是对已经存在的氨基酸物理化学性质和二级结构特性的扩展. Arg是Lys的替代体, Ser则对应Tyr, Val是疏水性氨基酸Leu, Ile和Met的补充[32~35].
最具吸引力的是六重简并的3个氨基酸Arg, Leu和Ser. 这些氨基酸在被引入后, 又由于核苷酸C在RNA世界的应用而扩展出各自的新四联码, 成为六重简并. 首先, Leu是在现代基因组中包括所有三界生物在内最丰富的氨基酸, Ser是真核生物第二丰富的氨基酸, Arg也是一个富有的氨基酸, 通常在细菌基因组中位于前10位. 其次, Leu在二重简并密码和四重简并之间最容易转换, 只需要通过简单U到C转换(UUR-CUR)即可, 这也说明Leu对于大多数蛋白质来说是用于当GC含量增加时维持蛋白质功能的完整性. 这些观察引出相应的假设: 这3个氨基酸的附加密码是为了当GC含量或AG含量增加时平衡富有氨基酸, 相应的密码分布按照平衡遗传密码的蛋白质多样性和蛋白质鲁棒性二等分. 这种平衡能力用于当编码序列突变发生时稳定蛋白质的氨基酸组成, 从而维护蛋白质结构的完整性.
(4) 遗传密码的最终拓展. 遗传密码的最终拓展是在DNA作为信息载体取代RNA使得信息载体具有更高的准确性和稳定性, 同时也产生了最为关键的从RNA到DNA的逆转录机制. 基于模板的DNA复制机制开辟了新的DNA-蛋白质-RNA世界. 很多新分子机制的进化包括DNA复制和修复、RNA的转录等, 使这个生物界里分子机制和细胞过程更趋于多元和完善. 同时当C和其脱氧衍生物分别作为结构模板加入RNA和DNA时, 标准遗传密码也就随之产生并被固定下来. 遗传密码本身得到新的补充并且编码能力有了很大提高. 组氨酸(His)和Glu立刻加入进来, 主要是由于它们具有相应的催化性质以及和原有的两个碱性氨基酸的相似性, Thr扩展了Ser的功能, 同时使蛋白质的结构增加了精细度, Ala同Ser相比具有类似的体积和尺度, 但其和Ser比具有很强的疏水性质[32,33]. 这些新引入的氨基酸在蛋白质结构和功能多样性上起到非常关键的作用. 不容怀疑的是Pro的最后加入, 它具有其他氨基酸所不具备的性质, 即通过特有的方式使蛋白质的骨架结构扭曲达到蛋白质结构的紧密折叠. 相应的扩展模式在AARS同样得到支持遗传密码扩展的假设, 除了3个六重简并的遗传密码外, 这次共有六组遗传密码最终被引入, 同时编码6个氨基酸. 这6个氨基酸的AARS分类按照G和I 的配对原则延伸而来. 例如AARS对于双重编码的氨基酸His(CAR)和Gln(CAY)的对应, Glu(GAR)和Asp(GAY)的对应等.
遗传密码的进化就是密码子的有序发生和合理分布, 这个分布的合理性一定经过一个复杂选择过程. 首先, 通过长时间的创造和优化, 使其在基因组核苷酸序列发生突变时对蛋白质的结构起到缓冲的作用; 第二, 密码子采取这样一种特殊的排布方式: 当DNA组成从AU富集区到GC富集区改变时, 氨基酸的分布倾向于从具有催化性质的氨基酸转到具有结构性质的氨基酸; 第三, 充分利用密码子第三位多变的优势(通常体现在R和Y之间的转换), 来改变编码氨基酸的物理化学性质, 致使在趋变半区里大约有15个氨基酸对第三个位置R和Y之间的转换呈现敏感.
(5) 分子机制与细胞过程的进化. 尽管分子机制与细胞过程的根本界限有时会很模糊, 但还是将它们分开: 前者强调物理性的相互作用、发生的空间和组分的存在, 后者强调化学反应的结果、发生的时间和过程. 从一方面讲, DNA的变异显然是细胞过程的产物, 遗传密码的发生和最终形成也是它的产物. 从另一方面讲, 密码子与氨基酸的关系影响到细胞的蛋白质组分的变化, 即分子机制的变化[21]. 比如, 如果在RNA世界需要产生多个拷贝的RNA分子, 一定需要一个分子机制来实现. 在现代生物世界里, 通常是由以DNA为模板的转录机制来完成, 但在RNA世界里没有RNA的复制, 多个RNA分子产生是由多聚酶和编辑体(Editosome)共同来完成的. 也许就是那个最原始的细胞机制. RNA世界的第二个分子机制发明可能是就剪接体, 这个分子机制在现代生物世界里仍然在发挥其重要的作用. 第三个分子机制也许是翻译体(Translatosome)的形成, 其用于直接进行蛋白质分子的加工, 这一分子机制是从原始的RNA世界到成熟的RNA世界再到现代生物世界里转折的重要标志. 在转折期里, 分子机制在蛋白质精确度的变化中不断完善和复杂, 直到DNA通过RNA和蛋白质的复合体引进到生命世界和逆转录体(Reverse Transcriptosome)的诞生. 在现代生物世界里, 生命体又最终发明了复制体(Replisome)、修复体(Repairosome)和转录体(Transcriptosome), 所有这些分子机制均以DNA为物质基础. 如果说翻译体是RNA世界终止的标志, 那么逆转录体也就意味着现代生物世界的诞生.
尽管遗传密码在现代生物世界里已经基本固定, 但新的分子机制和细胞过程还在不断地被创造, 它们不断地趋于完善和复杂, 新的物种也在不断地诞生. 代表生命的物种们也在不断的进化中走着不尽相同的道路, 要么维持鲁棒性(Robustness, 如细菌), 要么寻觅多样性(Diversity, 如节肢动物和植物), 要么追求复杂性(Complexity, 如脊椎动物). 很难相信遗传密码和它所编码的氨基酸就是铺垫所有这些道路的开创者和基石.
3 结束语
重排的密码表从根本上解释了DNA变异的规律与蛋白质编码序列(也就是氨基酸组成)之间的内在关系. 从这个关系里, 不仅找到了氨基酸物理化学性质与密码起源的关系, 也找到了可能的密码子进化和完善的途径, 并最终提出遗传密码的进化是与细胞的分子机制进化同步发生的. 理解遗传密码的起源和进化, 也就是理解生命的起源和进化.
尽管对于遗传密码的进化有很多假说, 本文也不想否定它们的合理性, 但是本文强调现代遗传密码的基本变化规律和生物学机制对人们自由遐想的限制. 事实不容许无限制地去推论, 人们需要的是寻求与遗传密码的起源和进化相关的事实或实验证据. 本研究提出遗传密码分步进化的机制, 不仅解释了遗传密码的分配机制是平衡氨基酸多样性和对DNA突变的鲁棒性, 也同时解释了AARS和遗传密码的共进化特征. 本研究组未来要揭示的是生命赖以存在的分子机制和细胞过程的起源和进化, 而不是单纯地探讨遗传密码的起源.
正如假说所预言: 如果AARS和tRNA联手的话, 遗传密码就有被重新排列的可能性. 但是如果现代氨基酸的存在不发生变化的话, 重排的结果可能还会是一样, 因为它们的理化性质是确定的. 也许在地球甚至地下的某一个角落里, 会发现某个氨基酸的根本缺失, 那时密码表显然会被改写, 但它的基本排列和对DNA突变压力的分解是不会改变的.
参考文献
1 Crick F H. The origin of the genetic code. J Mol Biol, 1968, 38: 367—379
2 Nirenberg M W, Matthaei J H. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA, 1961, 47: 1588—1602
3 Nirenberg M. Historical review: deciphering the genetic code-a personal account. Trends Biochem Sci, 2004, 29: 46—54
4 Knight R D, Freeland S J, Landweber L F. Selection, history and chemistry: the three faces of the genetic code. Trends Biochem Sci, 1999, 24: 241—247
5 Woese C R. Translation: in retrospect and prospect. RNA, 2001, 7: 1055—1067
6 Yu J. A content-centric organization of the genetic code. Geno Prot Bioinfo, 2007, 5: 1—6
7 Chargaff E. Structure and function of nucleic acids as cell constituents. Fed Proc, 1951, 10: 654—659
8 Chargaff E. How genetics got a chemical education. Ann N Y Acad Sci, 1979, 325: 344—360
9 Hu J, Zhao X, Zhang Z, et al. Compositional dynamics of guanine and cytosine content in prokaryotic genomes. Research in Microbiology, 2007, 158: 363—370
10 Amirnovin R. An analysis of the metabolic theory of the origin of the genetic code. J Mol Evol, 1997, 44: 473—476
11 Wong J T. A co-evolution theory of the genetic code. Proc Natl Acad Sci USA, 1975, 72: 1909—1912
12 Yockey H P. Origin of life on earth andShannon’s theory of communication. Comput Chem, 2000, 24: 105—123
13 Klipcan L, Safro M. Amino acid biogenesis, evolution of the genetic code and aminoacyl-tRNA synthetases. J Theo Biol, 2004, 228: 389—396
14 Davis B K. Evolution of the genetic code, Prog Biophys Mol Biol, 1999, 72: 157—243
15 Freeland S J, Knight R D, Landweber L F, et al. Early fixation of an optimal genetic code. Mol Biol Evol, 2000, 17: 511—518
16 Woese C R, Dugre D H, Dugre S A, et al. On the fundamental nature and evolution of the genetic code. Cold Spring Harbor Symp Quant Biol, 1966, 31: 723—736
17 Wu H L, Bagby S, Jean M H, et al. Evolution of the Genetic Triplet Code via Two Types of Doublet Codons. J Mol Evol, 2005, 61: 54—64
18 Delarue M. An asymmetric underlying rule in the assignment of codons: possible clue to a quick early evolution of the genetic code via successive binary choices. RNA, 2007, 13: 161—169
19 Houen G. Evolution of the genetic code: the nonsense, antisense, and antinonsense codes make no sense. BioSystems, 1999, 54: 39—46
20 Yu J. An evolutionary scenario for the origin of the genetic code. Communications of Chinese-American Chemical Society, 2007(Fall): 3—7
21 Xiao J, Yu J. A scenario on the stepwise evolution of the genetic code. Geno Prot Bioinfo, 2007, 5: 143—151
22 Cech T R, Bass B L. Biological catalysis by RNA. Annu Rev Biochem, 1986, 55: 599—629
23 Altman S, Baer M F, Bartkiewicz M, et al. Catalysis by the RNA subunit of RNase P. Gene, 1989, 82: 63—64
24 Noller H F, Hoffarth V, Zimniak L. Unusual resistance of peptidyl transferase to protein extraction procedures. Science, 1992, 256: 1416—1419
25 Szostak J W. Evolution ex vivo. Nature, 1993, 361: 119—120
26 Santoro S W, Joyce G F. Proc Natl Acad Sci USA, 1997, 94: 4262—4266
27 Turanov A A, Lobanov A V, Fomenko D E, et al. Genetic code supports targeted insertion of two amino acids by one codon. Science, 2009, 323: 259—261
28 Levy M, Miller S L. The stability of the RNA bases: implications for the origin of life. Proc Natl Acad Sci USA, 1998, 95: 7933—7938
29 Shapiro R. Prebiotic cytosine synthesis: a critical analysis and implications for the origin of life. Proc Natl Acad Sci USA, 1999, 96: 4396—4401
30 O'Donoghue P, Luthey-Schulten Z. On the Evolution of Structure in Aminoacyl-tRNA Synthetases. Microbiol Mol Biol Rev, 2003, 67: 550—573
31 Woese C R, Olsen G, Ibba M, et al. Aminoacyl-tRNA synthetases, the genetic code, and the evolutionary process. Microbiol Mol Biol Rev, 2000, 64: 202—236
32 Chothia C. The nature of the accessible and buried surfaces in proteins. J Mol Biol, 1975, 105: 1—12
33 Zamyatnin A A. Protein volume in solution. Prog Biophys Mol Biol, 1972, 24: 107—123
34 Chou P Y, Fasman G D. Prediction of protein conformation. Biochemistry, 1974, 13: 222—245
35 Chou P Y, Fasman G D. Empirical predictions of protein conformation. Annu Rev Biochem, 1978, 47: 251—276