本来还没这么快写进化专题的,但是有个朋友要我帮忙跑一下她的phy文件,看看能否生成树,我就试用了一下phyml这个软件,挺简单的。
一、下载并安装该软件
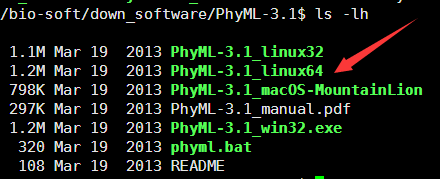
这是一个很简单的软件,我们直接下载它的二进制程序就可以直接使用啦,官网里面的压缩包里面有各种平台的二进制程序,我这里用linux64的。
- wget http://www.atgc-montpellier.fr/download/binaries/phyml/PhyML-3.1.zip
- unzip PhyML-3.1.zip
二.准备文件
它需要的phy格式的多重比对结果文件,一般是clustalW或者muscle比对的结果
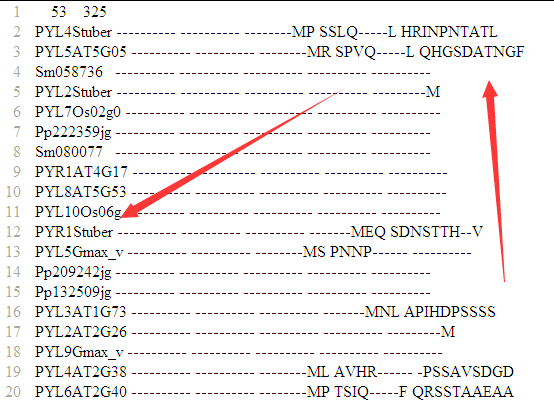
可以看到是53个蛋白,多重比对后的公共序列长度是325个氨基酸。
三.命令
./PhyML-3.1_linux64 -i test.phy -d aa -b 1000 -m LG -f m -v e -a e -o tlr
这些参数在运行的时候都会显示出来
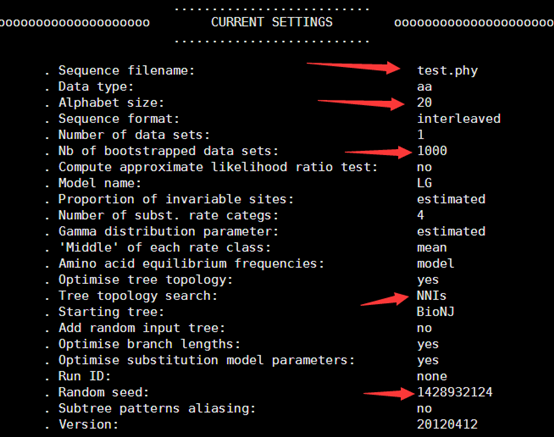
具体解释见: 使用 PhyML 构建进化树
四,输出文件
这个时间会很久,大家有心里准备!!!总共会输出四个文件,
test.phy_phyml_tree.txt : 最大似然法构建的进化树
test.phy_phyml_boot_stats.txt : bootstrap 的统计信息
test.phy_phyml_boot_trees.txt : bootstrap 树
test.phy_phyml_stats.txt : 程序运行的中的参数和结果统计
然后我们的那个test.phy_phyml_tree.txt 就可以用figtree等软件画图啦!!!
原文来自:http://www.bio-info-trainee.com/626.html


