软件的主页是
进入主页,简单看看软件介绍,这个软件还是蛮牛的,一个人在家里自己写出来的,当然,对于普通人来说,这个软件跟clustalW没什么区别,反正都是多序列比对啦!
我们下载适合我们平台的版本即可!
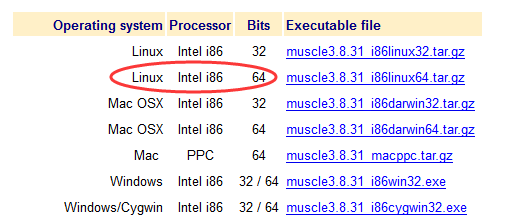
准备数据,我这里选择的是几个短小的蛋白
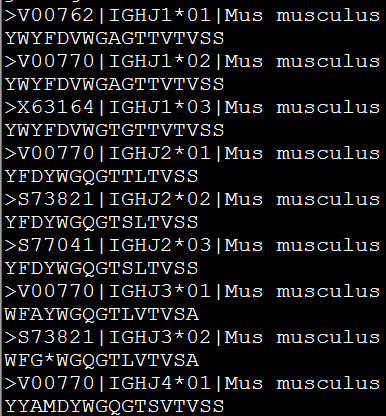
这里有两种比对方式,都是很简单的命令
一种是先比对,再生成树文件(树的格式是Newick format, )
muscle -in mouse_J.pro -out mouse_J.pro.a
muscle -maketree -in mouse_J.pro.a -out mouse_J.phy (这里有两种构建树的方式)
另外一种是比对成aln格式的数据,然后用其它软件(phyml或者phylip)来生出树文件
muscle -in mouse_J.pro -clwout seqs.aln
可以看到比对的效果还是蛮好的,是aln格式的比对文件,这个格式非常常用
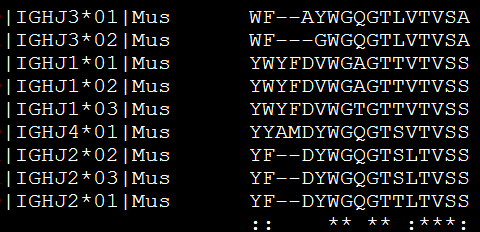
或者输出phy格式的比对文件,
muscle -in mouse_J.pro -physout seqs.phy
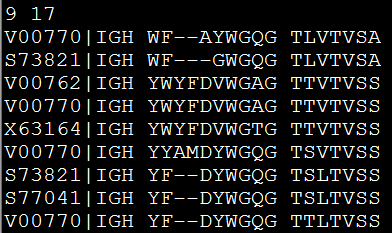
可以被phyml等软件识别,然后来构建进化树,https://www.plob.org/article/9891.html
原文来自:http://www.bio-info-trainee.com/659.html


