Gencode数据库是ENCODE计划的衍生品,也是由大名鼎鼎的sanger研究所负责整理和维护,主要记录了基因组的功能注释,比如基因组每条染色体上面有哪些编码蛋白的基因,哪些假基因,哪些lncRNA的基因,它们坐标是什么,基因上面的外显子内含子坐标是什么,UTR区域坐标是什么?我以前通常是在EBI的ENSEMBL的FTP服务器下载,后来才发现了这个Gencode数据库,现在以这个为金标准啦!
数据库文章:The GENCODE v7 catalog of human long noncoding RNAs, 链接是 http://genome.cshlp.org/content/22/9/1775.full
FTP地址:ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/ 可以下载该数据库的所有资料,而且整理的非常好,自己写脚本很容易处理得到自己想要的信息。
GENCODE最新版是v24,在linux系统里面用:
- wget -c -r -np -k -L -A "*metadata*" ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_24/
可以把所有metadata数据下载
检查里面的记录数:
- ls *gz |while read id;do (echo -n $id;echo -n " ";zcat $id |wc -l ) ;done
可以与官网的统计信息相对应: http://www.gencodegenes.org/stats.html
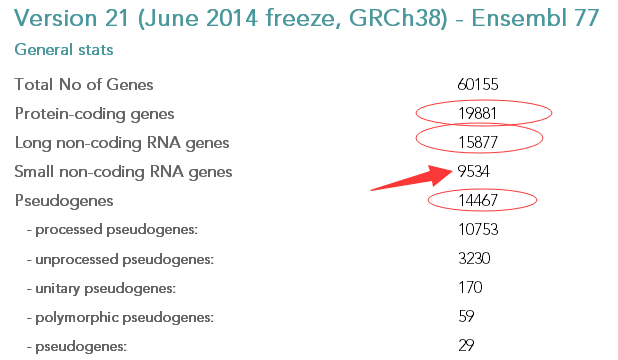
可以看到编码蛋白的基因的个数并不比lncRNA的基因多,甚至跟假基因也差不太多
首先,我们看看meta data信息,主要是该数据库与其它主流数据库的关系
- gencode.v24.metadata.Annotation_remark.gz 40879
- gencode.v24.metadata.EntrezGene.gz 170466
- gencode.v24.metadata.Exon_supporting_feature.gz 19193542
- gencode.v24.metadata.Gene_source.gz 66206
- gencode.v24.metadata.HGNC.gz 182831
- gencode.v24.metadata.PDB.gz 94547
- gencode.v24.metadata.PolyA_feature.gz 84652
- gencode.v24.metadata.Pubmed_id.gz 209094
- gencode.v24.metadata.RefSeq.gz 75365
- gencode.v24.metadata.Selenocysteine.gz 119
- gencode.v24.metadata.SwissProt.gz 45067
- gencode.v24.metadata.Transcript_source.gz 217202
- gencode.v24.metadata.Transcript_supporting_feature.gz 87375
- gencode.v24.metadata.TrEMBL.gz 61924
还可以下载所有的gtf文件:
- wget -c -r -np -nd -k -L -A “*gtf.gz” ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_24/
gtf文件特别重要,大家一定要抽两个小时时间好好理解,写一写脚本好好玩一玩这个文件,彻底吃透它!!!
还可以下载参考转录组及参考蛋白组,我这里还是拿hg19举例:
其实你有gtf文件,也可以直接从参考基因组序列里面提取这个参考转录组及参考蛋白组,就是通常是gtf2fasta,随便搜索一下,一大堆方法。
原文来自:http://www.bio-info-trainee.com/1781.html


