本来搞差异分析的工具和包就一大堆了,而且limma那个包已经非常完善了,我是不准备再讲这个的,正好有个同学问了一下这个包,我就随手测试了一下,顺便看看它跟limma有什么差异没有!手痒了就记录了测试流程!
学习一个包其实非常简单,就是找到包的官网看看说明书即可!说明书链接
samr这个包更简单,就一个函数SAM,但是根据分析数据的不同被包装成了两个函数,分别是处理高通量测序数据的SAMseq和处理芯片数据的samr,本次我只讲解芯片数据的处理,然后跟limma这个包做一个简单比较~
所以,我们只需要制作好数据,然后学会用samr这个函数即可!
我们还是利用CLL这个包的测试数据来讲解这个包的用法,首先也是制作表达矩阵和分组信息。
- suppressPackageStartupMessages(library(CLL))
- data(sCLLex)
- exprSet=exprs(sCLLex) ##sCLLex是依赖于CLL这个package的一个对象
- samples=sampleNames(sCLLex)
- pdata=pData(sCLLex)
- group_list=as.character(pdata[,2])
- group_list
- ## [1] "progres." "stable" "progres." "progres." "progres." "progres."
- ## [7] "stable" "stable" "progres." "stable" "progres." "stable"
- ## [13] "progres." "stable" "stable" "progres." "progres." "progres."
- ## [19] "progres." "progres." "progres." "stable"
- as.numeric(as.factor(group_list))
- ## [1] 1 2 1 1 1 1 2 2 1 2 1 2 1 2 2 1 1 1 1 1 1 2
这个表达矩阵exprSet和分组信息group_list就可以直接用来做差异分析啦~! 它的分组信息要求比较读取,需要1,1,1,2,2,2这样的向量,所以我用了as.numeric(as.factor(group_list)),具体见下面的代码!
- suppressPackageStartupMessages(library(samr))
- data=list(x=exprSet,y=as.numeric(as.factor(group_list)),
- geneid=as.character(1:nrow(exprSet)),
- genenames=rownames(exprSet),
- logged2=TRUE
- )
- samr.obj<-samr(data, resp.type="Two class unpaired", nperms=100)
这样其实已经OK啦,重点是如何调整这个函数的参数,以及如何理解这个函数返回的结果(samr.obj这个对象非常重要,关乎你能否真正用好samr)~
我这里的genenames其实是探针名,如果真正要做分析,可以修改,而且我的nperms次数为100,也可以修改,一般是1000.
除了直接应用它找差异基因外,它还有几个单独的函数
首先是对表达矩阵进行normalization
- x.norm par(mfrow=c(1,2))
- boxplot(exprSet, col = rainbow(exprSet),main="before normalization",las=2)
- boxplot(x.norm, col = rainbow(exprSet),main="after normalization",las=2)
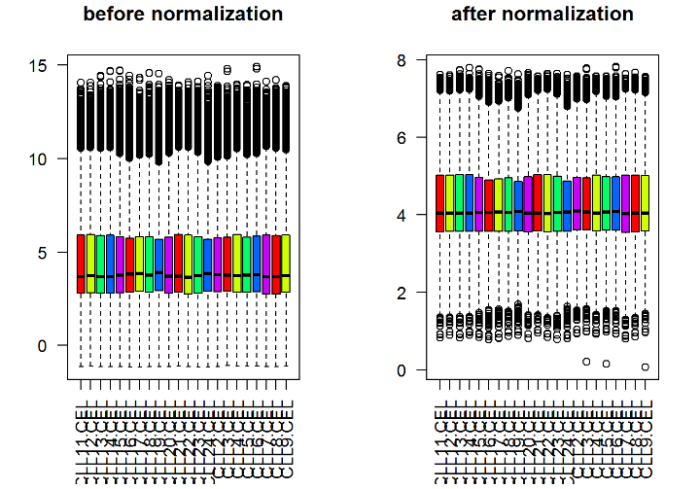
看图好像没什么区别
另外几个函数,我就不一一介绍了,大家可以自行探索。
* samr.plot(samr.obj, del, min.foldchange=0)
* samr.plot(samr.obj, del=.3)
* samr.assess.samplesize.obj
* samr.assess.samplesize.plot(samr.assess.samplesize.obj)
我们重点看看这个samr得到的差异与limma的差异区别在哪里
- ## 首先提取samr做差异分析检验的p值
- pv=samr.pvalues.from.perms(samr.obj$tt, samr.obj$ttstar)
- ## 然后提取limma包做差异分析检验的p值
- library(limma)
- design=model.matrix(~factor(sCLLex$Disease))
- fit=lmFit(sCLLex,design)
- fit=eBayes(fit)
- options(digits = 4)
- DEG_limma=topTable(fit,coef=2,adjust=\'BH\',n=Inf)
- pv_limma=DEG_limma$P.Value
- names(pv_limma)=rownames(DEG_limma)
- head(pv[sort(names(pv))])
- ## 100_g_at 1000_at 1001_at 1002_f_at 1003_s_at 1004_at
- ## 0.2531 0.4144 0.5671 0.5686 0.4687 0.6340
- head(pv_limma[sort(names(pv_limma))])
- ## 100_g_at 1000_at 1001_at 1002_f_at 1003_s_at 1004_at
- ## 0.2497 0.4312 0.5349 0.5498 0.4361 0.6473
- cor(pv[sort(names(pv))],pv_limma[sort(names(pv_limma))])
- ## [1] 0.9976
从数据上来看,没什么本质区别,而且相关系数高达0.9978.
所以结论是,没必要搞那么多的包,用limma就好了,甚至直接用t检验也是OK的
还有plot和summary也是可以直接作用于samr的结果samr.obj对象的。
原文来自:http://www.bio-info-trainee.com/1608.html


